聚合酶可以依照「DNA、RNA」區分成兩大類
又可按照「真核、原核」區分成兩大類
所以總共分為四象限
| 聚合酶命名 | 原核生物 | 真核生物 |
| RNA | 單一種類 | 羅馬數字 |
| DNA | 羅馬數字 | 希臘字母 |
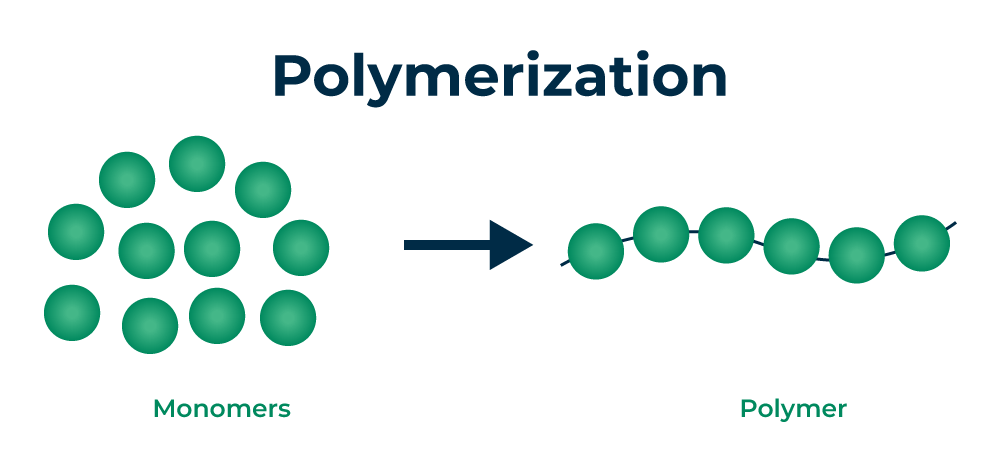
首先來看 polymerase 是什麼?
polymer 是「聚合物」,就是把很多單體(monomer)聚合起來形成的長鏈分子
polymerase 是「聚合酶」,是催化聚合反應的酶,就是具備組裝功能、生物界的自動組裝機
能把核苷酸nucleotide(ATCG)聚合成雙股DNA 的酵素,稱為DNA polymerase
能把核苷酸nucleotide(AUCG)聚合成單股RNA 的酵素,稱為RNA polymerase
因為原核沒有細胞核,真核才真正有細胞核
原核真核的兩套系統不同,用到的聚合酶也不一樣,這套命名系統看上去有點龐大、混亂
DNA 聚合酶(用途:複製DNA)
原核:羅馬數字
真核:希臘符號
| 生物類型 | 聚合酶名稱 | 主要功能 | 備註 | 來源基因 |
|---|---|---|---|---|
| 原核生物 | DNA Pol I | 移除 RNA primer 填補 DNA 缺口,修補損傷 | 具 5’→3’ 聚合、3’→5’ 校對、5’→3’ 外切酶活性 | polA |
| DNA Pol II | 損傷修復與錯誤修正 | 次要酵素,修復性較強 | polB | |
| DNA Pol III | 延長領先股和延遲股,沿 5′ → 3′ 方向合成新鏈 最主要的DNA複製酵素 | 高速高精準,具校對活性 | dnaE, dnaQ (核心次單元 α、ε) | |
| DNA Pol IV | 損傷容忍DNA合成(translesion synthesis) | 錯誤率高、無校正功能 | dinB | |
| DNA Pol V | 能跨越DNA損傷區域。在SOS 修復反應(SOS response)時,錯誤傾向的DNA合成 | 在DNA損傷時誘導表現,由RecA輔助活化。 | umuC、umuD | |
| 真核生物 | DNA Pol α | 啟動複製,與 primase 協作製造 RNA-DNA primer | 製作引子,作為起始模板 | POLA1 |
| DNA Pol β | 修復酶,在Base Excision Repair(BER) 中修補單鹼基缺損 | DNA修復(非同源末端接合) | POLB | |
| DNA Pol γ | 複製粒線體 DNA(mtDNA) | 位於粒線體(mitochondria) | POLG | |
| DNA Pol δ | 延伸 lagging strand | 具 3’→5’ 校對 | POLD1 | |
| DNA Pol ε | 延伸 leading strand | 主導主鏈複製 | POLE | |
| 其他 DNA Pol (κ、η、ζ 等) | DNA 修復、跨損傷複製 (translesion synthesis) | POLK, POLH, POLI |
RNA 聚合酶(用途:轉錄RNA)
原核:僅此一種,無編號
真核:羅馬數字
| 生物類型 | 聚合酶名稱 | 主要功能 | 備註 | 來源基因 |
|---|---|---|---|---|
| 原核生物 | RNA Pol(單一種) | 轉錄所有 RNA 包含 mRNA、tRNA、rRNA | 一種酶包辦所有轉錄 具多亞基 α₂ββ′σ | rpoA, rpoB, rpoC, rpoZ, rpoD |
| 真核生物 | RNA Pol I | 轉錄 rRNA(18S、28S、5.8S) | 位於核仁 nucleolus | POLR1A, POLR1B |
| RNA Pol II | 轉錄 mRNA、snRNA、miRNA | 基因表現主力 轉錄蛋白質編碼基因 | POLR2A, POLR2B | |
| RNA Pol III | 轉錄 tRNA、5S rRNA、小型 RNA | 位於核質(nucleoplasm)中,負責小 RNA | POLR3A, POLR3B | |
| RNA Pol IV | 植物特有,產生siRNA(小干擾RNA) | 僅見於植物,用於RNA沉默路徑 | NRPD1, NRPD2 | |
| RNA Pol V | 植物特有,協助RNA導引的DNA甲基化 | 與Pol IV共同調控表觀遺傳沉默 | NRPE1, NRPD2 |
真核生物通常要比原核生物複雜得多
原核是轉錄轉譯在同一個地方同時進行(自行煮火鍋,全部丟下去煮就好)
真核則是在不同地點不同時間進行(高級餐廳分成內場廚師與外場人員)
加上考慮到要進出細胞核核孔(進出門禁系統要刷卡),所以真核的工具箱裡面有更多種聚合酶
而DNA(雙股)複製過程也比轉錄RNA(單股)更為複雜,
因為DNA是遺傳物質,為了避免突變、力求高精準率
所以分工上更細更專業,DNA的工具種類也較多
⇒ 聚合酶數量:原核 < 真核 ;RNA < DNA
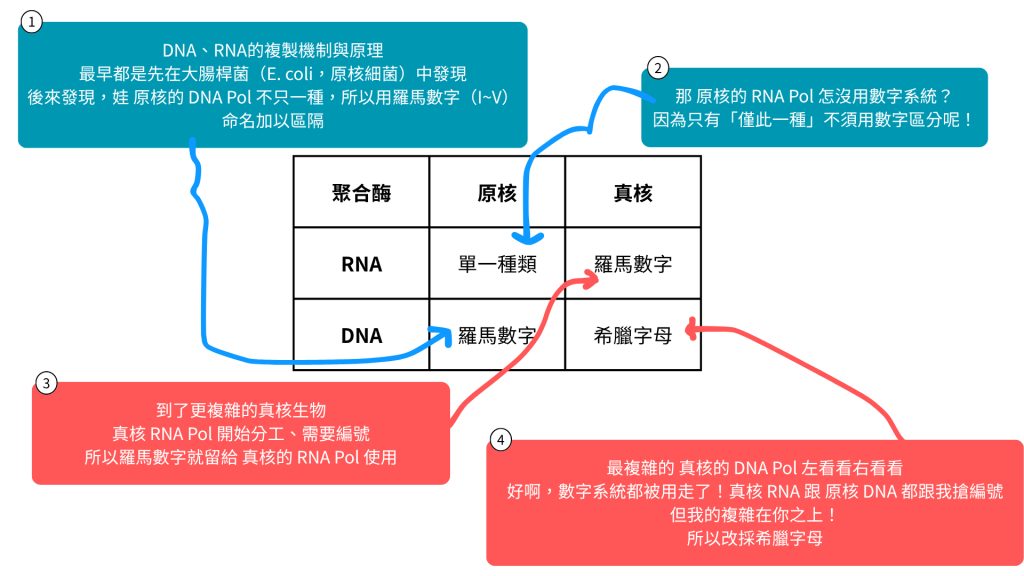
DNA、RNA的複製機制與原理,最早都是先在大腸桿菌(E. coli,原核細菌)中發現
後來發現,娃 原核的 DNA Pol 不只一種,所以用羅馬數字(I~V)命名加以區隔
那 原核的 RNA Pol 怎沒用數字系統?
因為只有「僅此一種」不須用數字區分呢!
到了更複雜的真核生物,真核 RNA Pol 開始分工、需要編號
所以羅馬數字就留給 真核的 RNA Pol 使用
最複雜的 真核的 DNA Pol 左看看右看看
好啊,數字系統都被用走了,真核 RNA Pol 跟 原核 DNA Pol 都跟我搶編號
但我的複雜在你之上!所以改採希臘字母
⇒ 命名系統:無編號(單一種) < 羅馬數字(2~5種)< 希臘字母(10種以上)
| 聚合酶分類 | 原核 (Prokaryote) | 真核 (Eukaryote) | 命名規則 |
|---|---|---|---|
| RNA polymerase | 1 種 (全功能) | I, II, III … (功能分工,I = rRNA, II = mRNA, III = tRNA/5S rRNA) | 原核:無編號 真核:羅馬數字 |
| DNA polymerase | I, II, III … (Pol I = 修復,Pol III = 主要複製) | α, δ, ε, γ, β… (依功能分工) | 原核:羅馬數字 真核:希臘字母 |
生活化比喻:
DNA polymerase 命名:像早期發現三種「螺絲起子」,就隨便叫「一號、二號、三號」,後來更多真核型號出現(十幾種),只好改用「A款、B款、C款」去標。
RNA polymerase 命名:在細菌工廠只有一台「印表機」負責印全部文件(rRNA、mRNA、tRNA);但真核公司比較複雜,有三台專門印不同部門文件,才叫「印表機 I、II、III」
老師上課常講:「RNA Pol II、DNA Pol I、DNA Pol III」這三個都具有複製模板、延長合成的功能,但不要看到直接拿來湊麻將「胡了」
因為一套是真核的、一套是細菌的
最經典也最容易考的是這三個
個人小抱怨
「Pol I II III」數字命名系統,為何要用羅馬數字… 超難辨認的
怎不用「Pol 1 2 3」哪!
「Pol II」跟「Pol ll(小寫L)」很像,而且筆記寫得靠近一點就眼花了「PolII」
(考慮私底下改用小寫字母「Pol ii」)
好在目前種類只有「I ~ V」數字小,
否則遇到更大數字「IX、XIV」還要想一下這是多少,一定更生氣
對凝血因子我就是在說你

發佈留言